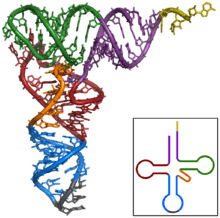
Structure tridimensionnelle d'un ARN régulateur (riboswitch).
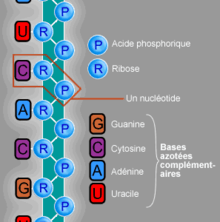
Structure moléculaire de l'ARN.
L'acide ribonucléique (ARN) est une molécule biologique présente chez pratiquement tous les êtres vivants, et aussi chez certains virus. L'ARN est une molécule très proche chimiquement de l'ADN et il est d'ailleurs en général synthétisé dans les cellules à partir d'une matrice d'ADN dont il est une copie. Les cellules vivantes utilisent en particulier l'ARN comme un support intermédiaire des gènes pour synthétiser les protéines dont elles ont besoin. L'ARN peut remplir de nombreuses autres fonctions et en particulier intervenir dans des réactions chimiques du métabolisme cellulaire.
Chimiquement, l'ARN est un polymère linéaire constitué d'un enchaînement de nucléotides. Chaque nucléotide contient un groupe phosphate, un sucre (le ribose) et une base nucléique, ou base azotée. Les nucléotides sont liés les uns aux autres par des liaisons phosphodiester. On trouve quatre bases nucléiques dans l'ARN : l'adénine, la guanine, la cytosine et l'uracile. L'ARN a de nombreuses similitudes avec l'ADN, avec cependant quelques différences importantes : d'un point de vue structurel, l'ARN contient des résidus de ribose là où l'ADN contient du désoxyribose, ce qui rend l'ARN chimiquement moins stable, tandis que la thymine de l'ADN y est remplacée par l'uracile, qui possède les mêmes propriétés d'appariement de base avec l'adénine. Sur le plan fonctionnel, l'ARN se trouve le plus souvent dans les cellules sous forme monocaténaire, c'est-à-dire de simple brin, tandis que l'ADN est présent sous forme de deux brins complémentaires formant une double-hélice. Enfin, les molécules d'ARN présentes dans les cellules sont plus courtes que l'ADN du génome, leur taille variant de quelques dizaines à quelques milliers de nucléotides, contre quelques millions à quelques milliards de nucléotides pour l'acide désoxyribonucléique (ADN).
Dans la cellule, l'ARN est produit par transcription à partir de l'ADN situé dans le noyau. L'ARN est donc une copie d'une région de l'un des brins de l'ADN. Les enzymes qui effectuent la copie ADN → ARN s'appellent des ARN polymérases. Les ARN ainsi produits peuvent avoir trois grands types de fonctions : ils peuvent être supports de l'information génétique d'un ou plusieurs gènes codant des protéines (on parle alors d'ARN messagers), ils peuvent adopter une structure secondaire et tertiaire stable et accomplir des fonctions catalytiques (par exemple l'ARN ribosomique), ils peuvent enfin servir de guide ou de matrice pour des fonctions catalytiques accomplies par des facteurs protéiques (ce qui est par exemple le cas des microARN).
Structure de l'ARN
Ribonucléotides
Structure chimique
Structure d'un brin d'ARN. Les atomes du ribose sont numérotés. La base représentée est l'uracile, qui n'est présente naturellement que dans l'ARN, à la place de la thymine, présente dans l'ADN.
L'ARN est un acide nucléique, c'est-à-dire une molécule constituée d'un enchaînement de nucléotides. Chaque nucléotide de l'ARN est constitué d'un pentose, le ribose, dont les atomes de carbone sont numérotés de 1′ à 5′, d'une base azotée, ou base nucléique, et d'un groupe phosphate. La base nucléique est reliée par un atome d'azote au carbone 1′ du ribose. Les nucléotides sont liés les uns aux autres par des groupes phosphate, par l'intermédiaire de liaisons phosphodiester au niveau des carbones 3′ et 5′. L'ARN possède quatre bases nucléiques différentes : l'adénine (notée A), l'uracile (noté U), la cytosine (notée C) et la guanine (notée G). La thymine de l'ADN est remplacée par l'uracile dans l'ARN. La différence entre ces deux bases est le remplacement d'un groupe méthyle en position 5 de la thymine par un atome d'hydrogène dans l'uracile. Cette modification de structure ne change pas les propriétés d'appariement avec l'adénine.
Adénine.
Guanine.
Cytosine.
Uracile.
Stéréochimie
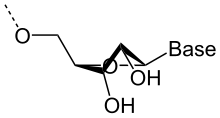
Conformation C2′-endo, observée dans les hélices de type B (ADN).
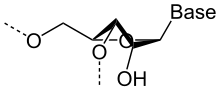
Conformation C3′-endo, observée dans les hélices de type A (ARN).
Sur le plan structurel, la présence d'un atome d'oxygène sur la position 2′ du ribose influence la conformation du cycle furanose du ribose. Cet hétérocycle à cinq atomes n'est pas plan, ce qui conduit à deux conformères principaux du sucre, appelés C2′-endo et C3′-endo. Dans l'ARN, qui comporte un atome d'oxygène en position 2′, la position C3′-endo est privilégiée, ce qui modifie profondément la structure des doubles hélices comportant des brins ARN. Ces duplex d'ARN forment une hélice de type A, différente de celle qui est observée de façon majoritaire dans l'ADN classique, qui est une hélice de type B, où le désoxyribose est en conformation C2′-endo.
Double hélice d'ARN
![Comparaison des structures de l'hélice B de l'ADN[7] et de l'hélice A de l'ARN[8]. Le grand sillon est fortement modifié.](https://wiki-gateway.eudic.net/wikipedia_fr/I/m/ABhelices.jpg)
Comparaison des structures de l'hélice B de l'ADN et de l'hélice A de l'ARN. Le grand sillon est fortement modifié.
L'hélice de type A qu'adopte l'ARN lorsqu'il forme un duplex a des propriétés géométriques assez différentes de celles de l'hélice de type B. Tout d'abord le nombre de paires de bases par tour d'hélice est de 11 au lieu de 10 pour l'ADN en forme B. Le plan des paires de bases n'est plus perpendiculaire à l'axe de l'hélice, mais forme un angle d'environ 75° avec celui-ci. Il en résulte un déplacement de l'axe de l'hélice qui ne passe plus par le centre de l'appariement des bases, mais à l'intérieur du grand sillon. Ceci induit une augmentation du diamètre de l'hélice qui passe d'environ 20 Å pour l'ADN en forme B à environ 26 Å pour l'ARN en forme A. Enfin, la géométrie des deux sillons est profondément affectée : le petit sillon devient très accessible, tandis que le grand sillon devient très profond, étroit et pincé. Ceci a un impact sur la manière dont l'ARN double brin peut interagir avec des protéines, car l'étroitesse du grand sillon est une barrière à l'accessibilité de ligands protéiques.
Structure in vivo
La plupart des ARN naturels sont présents sous forme monocaténaire (simple brin) dans la cellule, contrairement à l'ADN qui est sous forme d'un double brin apparié. Les brins d'ARN se replient le plus souvent sur eux-mêmes, formant une structure intramoléculaire qui peut être très stable et très compacte. La base de cette structure est la formation d'appariements internes, entre bases complémentaires (A avec U, G avec C et, parfois, G avec U). La description des appariements internes entre les bases d'un ARN s'appelle la structure secondaire. Cette structure secondaire peut être complétée par des interactions à distance qui définissent alors une structure tridimensionnelle ou structure tertiaire.
La formation de la structure des ARN est très souvent dépendante des conditions physicochimiques environnantes et en particulier de la présence, dans la solution, de cations divalents, comme l'ion magnésium Mg. Ces cations interagissent avec les groupes phosphate du squelette et stabilisent la structure, en particulier en faisant écran à la répulsion électrostatique entre les charges négatives de ces phosphates.
La structure tertiaire des ARN est à la base de la richesse de leurs fonctions et en particulier de leur capacité à catalyser des réactions chimiques (ribozymes).
Structure secondaire
Structure « en épingle à cheveux », ou tige-boucle, formée par une séquence palindromique sur l'ARN.
La structure secondaire d'un ARN est la description de l'ensemble des appariements internes au sein d'une molécule simple brin. Cet ensemble d'appariements induit une topologie particulière, composée de régions en hélice (tiges) et de régions non-appariées (boucles). Par extension, la structure secondaire recouvre également la description de cette topologie.
La formation de structures secondaires au sein d'un ARN simple brin résulte de l'e**stence de régions contenant des séquences palindromiques, qui peuvent s'apparier pour former localement une structure en double hélice. Par exemple, si l'ARN contient les deux séquences suivantes : --GUGCCACG------CGUGGCAC--, celles-ci forment une séquence palindromique, les nucléotides du second segment étant les complémentaires de ceux du premier, après inversion de leur sens de lecture ; ces deux segments peuvent alors s'apparier de manière antiparallèle pour former une région localement en duplex. La région entre les deux segments forme une « boucle » reliant les deux brins appariés, cet appariement formant une « tige ». On parle alors de structure en « épingle à cheveux », ou tige-boucle.
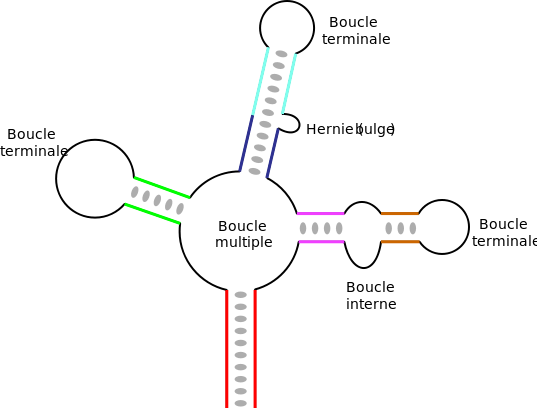
Dans des ARN de longueur plus importante, il peut e**ster des structures plus complexes qui résultent de l'appariement de plusieurs régions complémentaires ou palindromiques. En fonction de la manière dont sont « emboîtées » ces différentes régions, on obtient des éléments topologiques variés, avec des tiges ou régions appariées, et divers types de boucles :
les boucles terminales, situées à l'extrémité d'une tige ;
les boucles internes, qui connectent deux tiges ;
les boucles multiples, qui connectent trois tiges ou plus et constituent des points de branchement de la structure ;
les hernies (en anglais bulge), ou boucles latérales, qui sont sur un seul des deux brins d'une hélice. La continuité de l'hélice n'est en général pas affectée et les bases restent empilées de manière coa**ale, de part et d'autre de la hernie.
Il n'e**ste pas toujours une structure unique stable pour une séquence donnée et il arrive que certains ARN puissent adopter plusieurs conformations alternatives en fonction de la liaison d'un ligand (protéine, petite molécule…) ou des conditions physico-chimiques (force ionique, pH). On peut en général suivre la formation ou la fusion de la structure secondaire d'un ARN par des mesures spectroscopiques. Ainsi, par exemple, l'absorption dans l'ultraviolet des bases de l'ARN est plus importante à l'état déplié qu'à l'état replié (phénomène d'hyperchromicité).
Structure tertiaire
Exemple d'appariement non canonique : la paire G-A en cisaille (sheared). Celle-ci est présente par exemple dans la structure de l'ARN ribosomique 5S.
Appariements non canoniques
Au-delà de la topologie des boucles et des hélices composées de paires de bases standard, un ARN peut adopter une structure tridimensionnelle compacte, ou structure tertiaire, comme une protéine. À l'intérieur de cette structure, les hélices canoniques sont complétées par des appariements non canoniques, c'est-à-dire distincts des appariements classiques, de type Watson-Crick (A=U et G≡C) et bancals (wobble, G=U). On a observé une grande variété de ces appariements dans les structures tridimensionnelles d'ARN résolues par cristallographie aux rayons X ou par résonance magnétique nucléaire. On trouve par exemple des appariements Hoogsteen et des appariements « en cisaille » (sheared). Il e**ste également des interactions base-ribose, notamment avec l'hydroxyle 2', qui peut former des liaisons hydrogène. Une nomenclature systématique de toutes ces interactions a été proposée par Éric Westhof et ses collaborateurs. Plus de 150 types d'appariements ont été observés et ont été regroupés en douze grandes familles. Ces appariements non canoniques impliquent toujours des liaisons hydrogène entre les bases, qui sont coplanaires, comme dans les paires Watson-Crick.
Interactions à grande distance
Des appariements canoniques ou non canoniques peuvent intervenir entre des régions distantes de la structure secondaire, souvent localisées dans des boucles, ce qui permet de stabiliser un repliement compact de la structure.
Parmi ces interactions non canoniques à grande distance figurent :
les pseudonœuds, structures formées par l'interaction d'une boucle avec une région située à l'extérieur de la tige qui la délimite ;
les triplex de brin, qui surviennent lorsqu'une région simple brin vient s'insérer dans le grand sillon d'une région en hélice ;
les interactions tétraboucle-récepteur : interactions entre boucles hyperstables de quatre nucléotides (tétraboucles) et structures en duplex ou quasi-duplex.
Similitudes et différences entre l'ADN et l'ARN
Les principales différences entre les deux molécules sont que :
l'ARN a pour sucre le ribose là où l'ADN a du désoxyribose ;
la base uracile remplit dans l'ARN la fonction assurée par la thymine dans l'ADN ;
l'ARN e**ste généralement sous forme monocaténaire (simple brin), hormis chez quelques virus tels que les réovirus où il e**ste sous forme d'ARN double brin, tandis que l'ADN est double brin (bicaténaire) avec une structure en double hélice ;
l'ARN est court : de quelques dizaines à quelques milliers de nucléotides pour les ARN cellulaires (ARNm ou ARN structurés), contre quelques millions à quelques milliards (trois milliards chez l'Homme) dans l'ADN qui constitue le génome de la cellule.
Les trois premières différences donnent à l'ARN une stabilité bien moindre que celle de l'ADN :
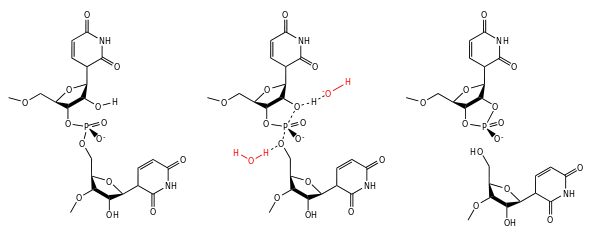
le ribose possède un groupe hydroxyle en position 2′, qui est absent dans le désoxyribose de l'ADN. Cette fonction 2′-OH a des incidences multiples sur la structure de l'ARN. Tout d'abord sur le plan chimique, cette fonction alcool rend l'ARN sensible à l'hydrolyse alcaline. La présence des deux oxygènes en cis sur les positions 2′ et 3′ rend possible la cyclisation du phosphate sur les positions 2′ et 3′, qui se produit très rapidement lorsqu'une base vient arracher le proton du 2′-OH. Cette cyclisation du nucléotide provoque une coupure de la chaîne ribose-phosphate et libère des extrémités 5′-OH et 2′, 3′ phosphate cyclique ;
l'uracile est moins coûteux énergétiquement à produire pour les organismes vivants que la thymine, puisqu'il nécessite une étape de synthèse de moins qu'est la méthylation par la thymidylate synthase. La présence de thymine dans l'ADN permet à la cellule de détecter des lésions spontanées de la cytosine qui est sensible à l'oxydation. La désamination spontanée de la cytosine en présence d'oxygène convertit cette dernière en uracile. La présence de désoxyuridine dans l'ADN est anormale, puisque le désoxyribonucléotide complémentaire de la désoxyadénosine est la thymidine. Grâce à cette distinction entre thymine et uracile au moyen d'un groupe méthyle, le système de réparation par excision de base peut détecter et corriger le défaut. Dans l'ARN, la désamination des cytosines produit des uraciles et n'est pas réparée. L'ARN ayant une durée de vie beaucoup plus courte que celle de l'ADN (de l'ordre de la minute), il est dégradé et recyclé ;
si un brin d'ARN monocaténaire est endommagé, la lésion n'est pas réparée et le dommage est irréversible ; en revanche, si un des deux brins d'ADN est endommagé, la cellule peut utiliser l'information portée par le brin complémentaire intact pour réparer la lésion.
D'un point de vue évolutif, certains éléments permettent de penser que l'ARN serait antérieur à l'ADN comme support de l'information génétique, ce qui expliquerait ses fonctions plus étendues et sa généralisation. L'ADN serait apparu plus tard et n'aurait supplanté l'ARN que pour le rôle de stockage à long terme, en raison de sa plus grande stabilité.
Synthèse de l'ARN à partir de l'ADN
La synthèse d'une molécule d'ARN à partir de l'ADN s'appelle la transcription. C'est un processus complexe qui fait intervenir une enzyme de la famille des ARN polymérases ainsi que des protéines associées. Les différentes étapes de cette synthèse sont l'initiation, l'élongation et la terminaison. Le processus de synthèse des ARN est sensiblement différent chez les organismes procaryotes et chez les cellules eucaryotes. Enfin, après la transcription proprement dite, l'ARN peut subir une série de modifications post-transcriptionnelles dans le cadre d'un processus de maturation au cours duquel sa séquence et sa structure chimique peuvent être modifiées (voir plus loin).
Initiation
Le démarrage de la transcription d'un ARN par une ARN polymérase s'effectue au niveau d'une séquence spécifique sur l'ADN, appelée promoteur. Ce promoteur comporte un ou plusieurs éléments de séquence conservés sur lesquels se fixent en général des protéines spécifiques, les facteurs de transcription. Juste en amont du site d'amorçage de la transcription, l'élément pro**mal est en général riche en nucléotides T et A, et est pour cette raison appelé boîte TATA chez les eucaryotes ou boîte de Pribnow chez les bactéries. Les facteurs de transcription favorisent le recrutement de l'ARN polymérase sur le promoteur et l'ouverture du duplex d'ADN. Il se forme alors ce qu'on appelle une bulle de transcription avec l'ADN ouvert, dont l'un des brins (la matrice) est hybridé avec l'ARN en cours de synthèse.
Élongation
![Vue en microscopie électronique des « arbres de Noël » ou « arbres de Miller » produits par la transcription d'un gène actif[29]. Le « tronc » de l'arbre est constitué de l'ADN et les « branches » sont les différentes molécules d'ARN en cours de synthèse. Celles-ci sont plus courtes vers le début de la région transcrite et plus longues vers la fin.](https://wiki-gateway.eudic.net/wikipedia_fr/I/m/Transcription_label_en.jpg)
Vue en microscopie électronique des « arbres de Noël » ou « arbres de Miller » produits par la transcription d'un gène actif. Le « tronc » de l'arbre est constitué de l'ADN et les « branches » sont les différentes molécules d'ARN en cours de synthèse. Celles-ci sont plus courtes vers le début de la région transcrite et plus longues vers la fin.
Une fois l'ARN polymérase fixée sur le promoteur et la bulle de transcription formée, elle synthétise les premiers nucléotides de manière statique sans quitter la séquence du promoteur. Les facteurs de transcription se détachent et l'ARN polymérase devient processive. Elle transcrit alors l'ARN dans le sens 5' vers 3', en utilisant l'un des deux brins de l'ADN comme matrice et des ribonucléotides triphosphates (ATP, GTP, CTP et UTP) comme précurseurs.
In vivo, chez Escherichia coli, la vitesse d'allongement de l'ARN polymérase est d'environ 50 à 90 nucléotides par seconde.
Terminaison
La terminaison de la transcription de l'ARN procède selon des mécanismes complètement différents chez les bactéries et chez les eucaryotes.
Chez les bactéries, le mécanisme principal de terminaison fait intervenir une structure particulière de l'ARN, le terminateur, composé d'une tige-boucle stable suivie d'une série de résidus d'uridine (U). Lorsque l'ARN polymérase synthétise cette séquence, le repliement de la tige d'ARN provoque une pause de la polymérase. L'ARN, qui n'est plus apparié à l'ADN matrice que par une série d'appariements A-U faibles, se détache, sans intervention d'autres facteurs protéiques. La terminaison peut aussi se faire via l'intervention d'un facteur protéique spécifique, le facteur Rho.
Chez les eucaryotes, la terminaison de la transcription par l'ARN polymérase II est couplée à la polyadénylation. Deux complexes protéiques, CPSF (en) et CStF (en) reconnaissent les signaux de polyadénylation (5′-AAUAAA-3′) et de coupure de l'ARN. Ils clivent l'ARN, induisent le détachement de la polymérase de l'ADN et recrutent la poly-A polymérase qui ajoute la queue poly(A) (voir plus bas).
Maturation
La maturation des ARN comprend un ensemble de modifications post-transcriptionnelles principalement observées chez les eucaryotes et jouent un rôle important dans le devenir de l'ARN maturé. Les principales modifications sont l'adjonction d'une coiffe en 5′, la polyadénylation en 3′, l'épissage, l'introduction de modifications chimiques au niveau de la base ou du ribose et enfin l'édition.
Coiffe
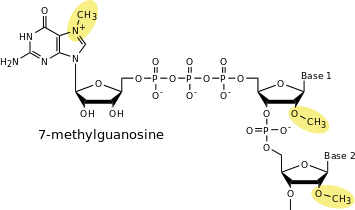
Structure de la coiffe en 5′ des ARN messagers.
La coiffe, ou 5′-cap en anglais, est un nucléotide modifié qui est ajouté à l'extrémité 5' de l'ARN messager dans les cellules d'eucaryotes. Elle se compose d'un résidu de guanosine méthylé lié par une liaison 5′-5′ triphosphate au premier nucléotide transcrit par l'ARN polymérase. Cette modification est introduite dans le noyau de la cellule, par l'action successive de plusieurs enzymes : polynucléotide 5'-phosphatase, ARN guanylyltransférase, méthyltransférases.
La coiffe joue plusieurs rôles : elle accroît la stabilité de l'ARN en le protégeant de la dégradation par des exonucléases 5′-3′ et permet également le recrutement de facteurs d'initiation de la traduction nécessaires à la fixation du ribosome sur les ARN messagers cellulaires. La coiffe est donc essentielle à la traduction de la plupart des ARNm.
Polyadénylation
La polyadénylation consiste en l'addition d'une extension à l'extrémité 3′ de l'ARN composée exclusivement de ribonucléotides de type adénosine (A). Pour cette raison, l'extension est appelée queue poly(A). Bien que composée de nucléotides standard, cette queue poly(A) est ajoutée de façon post-transcriptionnelle par une enzyme spécifique appelée poly(A) polymérase et n'est pas codée dans l'ADN génomique. La queue poly(A) est trouvée principalement à l'extrémité des ARN messagers. Chez les eucaryotes, la polyadénylation des ARNm est nécessaire à leur traduction par le ribosome et participe à leur stabilisation. La queue poly(A) est en particulier reconnue par la PABP (poly (A) binding protein, « protéine de liaison de la poly (A) »).
Chez les bactéries et dans certaines mitochondries, la polyadénylation des ARN est au contraire un signal de dégradation.
Épissage
Épissage des introns dans un ARN messager.
L'épissage est une modification post-transcriptionnelle qui consiste en l'élimination des introns et la suture des exons dans les ARN messagers et dans certains ARN structurés comme les ARNt. Présents chez les organismes eucaryotes, les introns sont des segments d'ARN qui sont codés dans le génome et transcrits dans l'ARN précurseur, mais qui sont éliminés du produit final. Dans la plupart des cas, ce processus fait intervenir une machinerie spécifique complexe appelée le splicéosome. L'épissage se produit dans le noyau des cellules eucaryotes, avant l'export de l'ARN maturé vers le cytoplasme.
Nucléotides modifiés
Après leur transcription par l'ARN polymérase, certains ARN subissent des modifications chimiques sous l'action d'enzymes spécifiques. Les principaux ARN subissant des modifications sont les ARN de transfert et les ARN ribosomiques. On peut également considérer que les méthylations intervenant dans la synthèse de la coiffe sont des modifications de nucléotides particulières. Dans le cas général, les modifications peuvent porter soit sur la base, soit sur le ribose. Les principales modifications rencontrées sont :
sur le ribose : des O-méthylations de la position 2′-OH ;
sur la base : l'isomérisation des uridines qui donne des pseudouridines, des méthylations, soit sur des atomes de carbone (ribothymidine), soit sur des atomes d'azote (7-méthylguanosine...), une réduction, qui transforme les uridines en dihydrouridines, des thiolations, des modifications plus complexes (prénylation, thréonyl-carbamoylation...).
l'isomérisation des uridines qui donne des pseudouridines,
des méthylations, soit sur des atomes de carbone (ribothymidine), soit sur des atomes d'azote (7-méthylguanosine...),
une réduction, qui transforme les uridines en dihydrouridines,
des thiolations,
des modifications plus complexes (prénylation, thréonyl-carbamoylation...).
Dans les ARN de transfert, l'introduction de nucléotides modifiés contribue à augmenter la stabilité des molécules.
Édition
L'édition des ARN consiste en une modification de la séquence de l'acide ribonucléique, postérieure à la transcription par l'ARN polymérase. À l'issue du processus d'édition, la séquence de l'ARN est donc différente de celle de l'ADN. Les changements opérés peuvent être la modification d'une base, la substitution d'une base ou encore l'ajout d'une ou plusieurs bases. Ces modifications sont effectuées par des enzymes qui agissent sur l'ARN, comme les cytidine désaminases, qui transforment chimiquement les résidus de cytidine en uridine.
Fonction dans la cellule
Dans les cellules, les ARN remplissent quatre rôles distincts et complémentaires :
support temporaire de l'information génétique : c'est l'ARN messager qui remplit ce rôle, il est utilisé par la cellule pour transmettre l'information correspondant à un gène donné à l'extérieur du noyau, puis pour synthétiser des protéines à partir de ces informations ;
catalyseur enzymatique : comme les protéines, les ARN peuvent se replier en trois dimensions pour former des structures complexes. Ces structures permettent à certains ARN de se comporter comme des enzymes, on parle alors de ribozyme. Le ribosome, la ribonucléase P et certains introns sont des ribozymes. Il e**ste des arguments indirects indiquant que la machinerie d'épissage des ARN messagers (le splicéosome) est également aussi un ribozyme, même si la démonstration formelle n'en a pas encore été apportée ;
guide pour des enzymes : certains ARN sont utilisés comme cofacteurs par des protéines pour permettre leur ciblage vers des séquences spécifiques. Parmi ceux-ci, on peut citer les petits ARN nucléolaires (snoARN), qui guident les enzymes de modification de l'ARN ribosomique, l'ARN télomérique, qui est un cofacteur de la télomérase, l'enzyme qui fabrique les extrémités des chromosomes, ou encore les ARN interférents ;
régulateurs de l'expression génétique : certains ARN non codants jouent un rôle dans la répression de l'expression de certains gènes ou groupes de gènes. C'est le cas par exemple des ARN antisens qui s'apparient à un ARN cible et en bloquent la traduction par le ribosome.
Une classe particulière d'ARN, les ARN de transfert, se trouve à l'interface de plusieurs de ces fonctions en guidant les acides aminés lors de la traduction.
Enfin, le génome de certains virus est exclusivement constitué d'ARN et non d'ADN. C'est en particulier le cas des virus de la grippe, du SIDA, de l'hépatite C, de la poliomyélite ou encore du virus Ebola. Suivant les cas, la réplication de ces virus peut passer par un intermédiaire ADN (rétrovirus), mais peut aussi se faire directement d'ARN en ARN.
L'ARN est donc une molécule très polyvalente, ce qui a conduit Walter Gilbert, co-inventeur du séquençage de l'ADN, à proposer en 1986 une hypothèse selon laquelle l'ARN serait la plus ancienne de toutes les macromolécules biologiques. Cette théorie, dite RNA world hypothesis (« hypothèse du monde à ARN »), permet de s'affranchir d'un paradoxe de l'œuf et de la poule qui survient lorsqu'on cherche à savoir qui des protéines (catalyseurs) et de l'ADN (information génétique) sont apparus en premier. Dans ce modèle, l'ARN, capable de combiner à la fois les deux types de fonctions, serait le précurseur universel.
ARN messagers
L'information génétique contenue au sein de l'ADN n'est pas utilisée directement par la cellule pour synthétiser des protéines. Celle-ci utilise pour cela des copies transitoires de l'information génétique que sont les ARN messagers ou ARNm. Chaque ARN messager porte un ou, parfois, plusieurs cistrons, c'est-à-dire les instructions pour former une seule protéine. Il correspond donc à la copie d'un seul des gènes du génome (on parle alors d'ARNm monocistronique) ou parfois de quelques-uns (ARNm polycistronique).
L'ARN messager ne contient la copie que d'un seul des deux brins de l'ADN, celui qui est codant, et non la séquence complémentaire. Par rapport à la séquence du gène contenue dans l'ADN du génome, celle de l'ARNm correspondant peut contenir des modifications, en particulier dues à l'épissage (voir plus haut) qui élimine les régions non codantes. L'ARN messager synthétisé dans le noyau de la cellule est exporté dans le cytoplasme pour être traduit en protéine. Contrairement à l'ADN, qui est une molécule pérenne, présente pendant toute la vie de la cellule, les ARN messagers ont une durée de vie limitée, de quelques minutes à quelques heures, après quoi ils sont dégradés et recyclés.

Un ARN messager comporte trois régions distinctes : une région 5′ non traduite dite 5′-UTR, située en amont du ou des cistrons qu'il porte ; une région codante correspondant à ce ou à ces cistrons ; et enfin, une région 3′ non traduite dite 3′-UTR. Les ARN messagers sont traduits en protéines par les ribosomes. La région 5′ non traduite contient en général les signaux de traduction permettant le recrutement du ribosome sur le cistron. Le processus de traduction fait également intervenir les ARN de transfert qui apportent au ribosome les acides aminés nécessaires à la biosynthèse des protéines. Au sein du ribosome, par leur anticodon, les ARNt s'apparient successivement aux triplets de bases, ou codons, de la séquence de l'ARNm. Lorsque l'appariement codon-anticodon est correct, le ribosome ajoute l'acide aminé porté par l'ARNt à la protéine en cours de synthèse. Les correspondances entre codons et acides aminés constituent le code génétique.
La fonction des ARN messagers est multiple. Ils permettent d'une part de préserver la matrice d'ADN originale, qui n'est pas directement utilisée pour la traduction, la cellule ne travaillant que sur la copie qu'est l'ARNm. L'e**stence d'ARN messagers offre surtout à la cellule un mécanisme crucial de régulation du cycle de production des protéines à partir du génome. Le besoin cellulaire en telle ou telle protéine peut varier en fonction de l'environnement, du type de cellule, du stade de développement. La synthèse protéique doit donc être activée ou arrêtée en fonction des conditions cellulaires. La régulation de la transcription de l'ADN en l'ARNm répond à cette nécessité et est contrôlée par des facteurs de transcription spécifiques agissant sur les promoteurs des gènes cibles. Lorsque la quantité d'une protéine donnée est suffisante, la transcription d'ARNm est inhibée, celui-ci est progressivement dégradé et la production protéique cesse. Il est donc important que l'ARNm soit une molécule transitoire, afin de pouvoir réaliser cette régulation essentielle.
ARN de transfert
Vue tridimensionnelle de l'ARN de transfert pour la phénylalanine chez la levure (PDB 1EHZ) : - jaune : site de l'acide aminé ; - violet : tige accepteur ; - vert : boucle TΨC ; - rouge : branche D ; - orange : boucle variable ; - bleu : boucle de l'anticodon ; - gris : anticodon.
Les ARN de transfert, ou ARNt, sont de courts ARN, longs d'environ 70 à 100 ribonucléotides, impliqués dans l'adressage des acides aminés vers les ribosomes lors de la traduction.
Les ARN de transfert ont une structure caractéristique en feuille de trèfle, composée de quatre tiges appariées. L'une de ces tiges est terminée par une boucle qui contient l'anticodon, le triplet de nucléotides qui s'apparie au codon lors de la traduction d'un ARNm par le ribosome. À l'autre extrémité, l'ARNt porte l'acide aminé correspondant attaché par une liaison ester à son extrémité 3′-OH. Cette estérification est catalysée par des enzymes spécifiques, les aminoacyl-ARNt synthétases. En trois dimensions, la structure en feuille de trèfle se replie en « L », avec l'anticodon à une extrémité et l'acide aminé estérifié à l'autre extrémité.
Toutes les cellules vivantes contiennent un ensemble d'ARNt différents portant les différents acides aminés et capable de lire les différents codons.
Les ARN de transfert sont parfois désignés comme des « adaptateurs » entre la séquence génétique et la séquence protéique. C'est Francis Crick qui a proposé l'e**stence de ces adaptateurs, avant même leur découverte en 1958.
ARN catalytiques ou ribozymes
Structure de la ribonucléase P, une enzyme présente dans toutes les cellules vivantes et dont l'activité enzymatique est portée par un ARN.
La découverte d'ARN possédant des capacités catalytiques a été faite dans les années 1980, en particulier par l'équipe de Thomas Cech, qui travaillait sur les introns du gène de l'ARN ribosomique du protozoaire cilié Tetrahymena, et celle de Sidney Altman, qui étudiait la ribonucléase P, l'enzyme de maturation de l'ARNt. Cech et Altman ont été récompensés par le prix Nobel de chimie en **** pour cette découverte.
Dans ces deux cas, l'ARN seul est capable de catalyser une réaction de clivage (coupure) ou de transestérification spécifique en l'absence de protéine. Ces ARN catalytiques ont été appelés ribozymes, car ce sont des enzymes constituées d'acide ribonucléique. Dans le cas de l'intron de Tetrahymena, il s'agit d'un auto-épissage, l'intron étant son propre substrat, tandis que la ribonucléase P est une enzyme agissant en trans, sur des substrats multiples.
Structure du ribozyme en tête de marteau présent dans le génome d'un viroïde infectant le plant d'avocat.
Depuis ces découvertes initiales, d'autres ribozymes naturels ont été identifiés :
les ARN de viroïdes ou de virus satellites (virusoïdes) qui sont capables de se cliver eux-mêmes ;
il e**ste aujourd'hui des arguments très forts, basés sur la résolution de sa structure 3D, pour affirmer que le ribosome, le complexe de ribonucléoprotéines responsable de la traduction de l'ARNm en protéines, est lui-même un ribozyme. Les deux sites actifs du ribosome, le centre de décodage sur la petite sous-unité et le centre peptidyltransférase qui forme les liaisons peptidiques, sont en effet exclusivement composés de segments d'ARN ribosomique ;
le splicéosome, qui catalyse l'épissage des ARNm cytoplasmiques des eucaryotes, est probablement aussi un ribozyme ;
certains riboswitchs, qui sont des régions régulatrices structurées portées par des ARN messagers, ont une activité catalytique de coupure en présence d'un ligand ;
il e**ste enfin des ribozymes synthétiques, qui ont été isolés par des méthodes d'évolution in vitro, comme la technique SELEX. On a ainsi pu isoler des ARN catalytiques synthétiques capables de catalyser une grande variété de réactions chimiques et de fixer des ligands très divers, ce qui a été interprété comme un argument en faveur de l'hypothèse du monde à ARN. De tels ARN synthétiques sont parfois appelés des aptamères, puisqu'ils sont « aptes » à réaliser une tâche donnée.
De manière générale, dans tous ces ribozymes, c'est leur repliement spécifique qui leur permet d'effecteur la reconnaissance de leur substrat et la catalyse, comme dans le cas des enzymes protéiques.
ARN guides
Les ARN guides sont des ARN qui s'associent à des enzymes protéiques et servent à en guider l'action sur des ARN ou des ADN de séquence complémentaire. L'ARN guide s'apparie à l'acide nucléique substrat et permet de cibler l'activité de l'enzyme. On a identifié plusieurs types :
les petits ARN nucléolaires, ou snoARN : dans le nucléole des cellules eucaryotes, ils dirigent l'action d'enzymes de modification de l'ARN ribosomique, en particulier les 2′-O-méthylations par les snoARN C/D et les pseudouridylations par les snoARN H/ACA. Ce mécanisme permet à la cellule de modifier spécifiquement de multiples positions de l'ARNr, avec une seule enzyme en utilisant différents snoARN comme guides. Les snoARN sont souvent codés par des séquences introniques ;
les microARN sont également des ARN guides qui interviennent dans le processus d'interférence par ARN : associés à un complexe protéique appelé RISC (RNA induced silencing complex), ces petits ARN provoquent soit une dégradation de l'ARNm cible auquel ils s'apparient, soit une répression de sa traduction ;
TERC (telomerase RNA component), la sous-unité ARN de la télomérase : cet ARN structuré est associé à la transcriptase inverse qui synthétise les télomères, extrémités des chromosomes. Il contient une séquence qui sert de substrat à la télomérase pour synthétiser l'ADN télomérique de séquence complémentaire. Il guide donc l'activité de l'enzyme, mais en servant de matrice, plutôt qu'en formant un appariement avec le substrat ;
les lincARN, présents chez les mammifères, sont de grands ARN intergéniques non codants mais transcrits comme les ARNm par l'ARN polymérase II. Leur longueur leur permet d'adopter une structure tridimensionnelle complexe. Ces structures permettent leur interaction avec différent cofacteurs transcriptionnels tel que hnRNP-K ou PRC2 (principalement des inhibiteurs de la transcription). Ces complexes sont ensuite guidés grâce aux lincARN sur les séquences régulatrices des gènes afin d'inhiber leur expression. La liaison des lincARN avec l'ADN impliquerait un appariement des bases ARN avec les bases ADN correspondantes après désappariement de la double hélice d'ADN, voire la formation de triple hélice ADN-ADN-ARN.
ARN régulateurs
Chatte au pelage tricolore, dit écaille de tortue, lié à une inactivation du chromosome X par l'ARN **. Seules les femelles XX présentent ce pelage mélangé.
Certains ARN jouent un rôle de régulateurs directs de l'expression génétique. C'est en particulier le cas d'ARN non codants possédant des régions complémentaires d'ARN messagers cellulaires et qui peuvent donc s'y apparier pour former localement un double brin d'ARN. Ces ARN antisens peuvent être issus du même locus génétique que leur ARN cible, par transcription du brin complémentaire, on parle alors d'ARN cis-régulateurs. Ils peuvent aussi être issus de la transcription d'une autre région du génome, ce sont alors des ARN trans-régulateurs.
L'appariement de l'ARN régulateur avec son ARN messager cible peut agir sur la capacité de ce dernier à être traduit par le ribosome ou sur sa stabilité, ce qui aboutit à une régulation de la traduction du ou des gènes portés par l'ARN messager. Chez les bactéries, il e**ste ainsi de nombreux exemples d'ARN antisens cis- ou trans-régulateurs qui bloquent le site de démarrage de la traduction. Par exemple, le gène codant la porine OmpF est régulé par un ARN antisens appelé MicF.
Chez les eucaryotes, il e**ste aussi de grands ARN régulateurs, qui interviennent dans des processus de régulation épigénétique. L'exemple le mieux connu est celui de l'ARN ** chez les mammifères. Celui-ci inactive non pas un gène, mais un chromosome entier. **st recouvre l'un des deux chromosomes X de chaque cellule chez les individus femelles qui devient ainsi inactif. Un seul des deux chromosomes de la paire XX est ainsi actif, ce qui permet d'avoir le même taux d'expression des gènes portés par le chromosome X que chez les individus mâles, qui n'en ont qu'un. L'inactivation du X est un processus aléatoire, ce qui peut conduire à l'expression de différents phénotypes par différentes cellules, chez la même femelle. C'est par exemple le cas pour la couleur du pelage chez les chattes.
Utilisations thérapeutiques et biotechnologiques
L'ARN est utilisé aujourd'hui dans un certain nombre d'applications en biologie moléculaire, en particulier grâce au processus d'interférence par ARN, qui consiste en l'introduction dans des cellules eucaryotes de courts fragments d'ARN double-brin appelés « petits ARN interférents ». Longs d'une vingtaine de paires de bases, ces petits ARN interférents (pARNi) sont utilisés par une machinerie cellulaire capable de dégrader les ARNm de manière spécifique. Seuls les ARNm contenant une séquence correspondant à celle du pARNi sont dégradés, ce qui permet de diminuer sélectivement l'expression d'une protéine donnée. Cette approche technologique est beaucoup plus simple et rapide que l'établissement de lignées murines inactivées (knock-out) et s'appelle un knock-down.
Des essais d'utilisation de cette technique à des fins thérapeutiques sont envisagés, par exemple en ciblant des gènes viraux pour lutter contre des infections, ou des oncogènes, dans le cas de cancers. Ils nécessitent cependant de stabiliser les petits ARN interférents (pARNi) pour éviter leur dégradation par des ribonucléases et de cibler leur action vers les cellules concernées.
Historique
Les acides nucléiques ont été découverts en 1868 par Friedrich Miescher. Miescher appela la nouvelle substance « nucléine » car elle se trouvait dans le noyau des cellules. La présence d'acides nucléiques dans le cytoplasme de la levure fut identifiée en 1939 et leur nature ribonucléique fut établie, contrairement aux chromosomes qui contenaient de l'ADN avec des désoxyriboses.
Vers 1940, le biologiste belge Jean Brachet étudie des molécules jusque-là peu caractérisées, que l'on appelle encore à l'époque les "acides thymonucléiques et zymonucléiques" (respectivement l'ADN et l'ARN). Il découvre que l'acide thymonucléique est un composant des chromosomes et qu'il est synthétisé lorsque les cellules se divisent après la fécondation. Il met en évidence l'e**stence d'acides zymonucléiques (ARN) dans tous les types cellulaires: dans le noyau, le nucléole et le cytoplasme de toutes les cellules (alors que l'on pensait à l'époque que ces molécules étaient caractéristiques des cellules végétales et des eucaryotes inférieurs tels que les levures). Enfin, il montre que ces acides sont particulièrement abondants dans les cellules (plus particulièrement dans l'ergastoplasme) qui sont très actives en termes de synthèse protéique. Les bases fondamentales de la biologie moléculaire étaient établies. Nous étions en 1940. Dans l'après-guerre, Brachet est rejoint par le biologiste moléculaire belge Raymond Jeener qui participera activement aux recherches sur le rôle de l'ARN dans la biosynthèse des protéines.
À la fin des années 1950, Severo Ochoa parvint à synthétiser in vitro des molécules d'ARN au moyen d'une enzyme spécifique, la polynucléotide phosphorylase, ce qui permit l'étude des propriétés chimiques et physiques de l'ARN.
Le rôle de l'ARN comme « messager » intermédiaire entre l'information génétique contenue dans l'ADN et les protéines fut proposé en 1960 par Jacques Monod et François Jacob à la suite d'une discussion avec Sydney Brenner et Francis Crick. La démonstration de l'e**stence de l'ARN messager a été faite par François Gros. Ensuite, le déchiffrage du code génétique a été réalisé par Marshall Nirenberg dans la première moitié des années 1960. Il utilisa pour cela des ARN synthétiques de séquence nucléotidique connue dont il étudia les propriétés de codage.
Les ribosomes furent observés pour la première fois par le biologiste belge Albert Claude au début des années 1940. Par des techniques de fractionnement subcellulaire et de microscopie électronique, il mit en évidence des « petites particules » de nature ribonucléoprotéique, présentes dans tous les types de cellules vivantes. Il les baptisa « microsomes », plus tard renommés ribosomes.
La structure secondaire des ARNt a été établie par Robert Holley, qui est parvenu à purifier et à analyser la séquence de l'ARNt spécifique de l'alanine en 19**. Ce fut un progrès majeur dans la compréhension du déchiffrage du message génétique porté par les ARN messagers. La structure tridimensionnelle d'un ARNt a été résolue en 1974, indépendamment, par les équipes de Aaron Klug et Alexander Rich, montrant pour la première fois la structure complexe d'un ARN. L'e**stence de propriétés catalytiques des ARN a été établie indépendamment par Sidney Altman et Tom Cech en 1982, sur la ribonucléase P d'une part et sur les introns autoépissables d'autre part. La résolution de la structure des sous-unités individuelles du ribosome en 2000 par les équipes de Tom Steitz, Ada Yonath et Venki Ramakrishnan, puis celle du ribosome entier par l'équipe de Harry Noller en 2001, constituèrent un progrès essentiel dans la compréhension du mécanisme central de la biologie qu'est la traduction des ARNm en protéines. De plus, cela permit de montrer, entre autres, que le ribosome était aussi un ribozyme.
Durant les années 1970, Timothy Leary, dans son ouvrage La Politique de l'Extase, verra dans l'ARN la promesse d'une future modification de la conscience (possiblement via de nouvelles drogues, et/ou d'exercices spirituels), dont il serait une composante agrandissant les capacités d'apprentissage de celui qui se livrerait à de telles expériences.
Hypothèse du monde à ARN
L'hypothèse du monde à ARN est une hypothèse suivant laquelle l'ARN serait le précurseur de toutes les macromolécules biologiques et particulièrement de l'ADN et des protéines qui auraient permis dans un environnement abiotique (caractérisé par une chimie prébiotique en partie hypothétique) l'apparition de premières cellules vivantes, c'est-à-dire, formant un compartiment, et comportant de l'information et des sous-systèmes métaboliques.
Dans le cadre de l'étude des origines de la vie, cette hypothèse permet une explication de l'apparition des différentes fonctions biologiques via la constitution de certains blocs biomoléculaires à partir d'intermédiaires prébiotique plausibles et de molécules basées sur le carbone. Il a été démontré que des précurseurs plausibles des ribonucléotides, des acides aminés et des lipides peuvent tous être obtenus par homologation réductrice du cyanure d'hydrogène et de certains de ses dérivés. Chacun des sous-systèmes cellulaires connus pourrait donc être expliqué par la chimie du carbone, avec des réaction catalysées par la lumière ultraviolette a priori très présente avant l'apparition de la couche d'ozone, à partir du sulfure d'hydrogène comme agent réducteur. Le cycle photoréducteur pourrait lui-même être accéléré par le cuivre [Cu(I)-Cu(II)].

 词典释义:
词典释义:



![Comparaison des structures de l'hélice B de l'ADN[7] et de l'hélice A de l'ARN[8]. Le grand sillon est fortement modifié.](https://wiki-gateway.eudic.net/wikipedia_fr/I/m/ABhelices.jpg)


![Vue en microscopie électronique des « arbres de Noël » ou « arbres de Miller » produits par la transcription d'un gène actif[29]. Le « tronc » de l'arbre est constitué de l'ADN et les « branches » sont les différentes molécules d'ARN en cours de synthèse. Celles-ci sont plus courtes vers le début de la région transcrite et plus longues vers la fin.](https://wiki-gateway.eudic.net/wikipedia_fr/I/m/Transcription_label_en.jpg)